Dalla linea allo spazio: lezioni di architettura dal genoma umano
Per anni abbiamo considerato il DNA solo ed esclusivamente nella sua struttura lineare. Negli ultimi anni però la biologia molecolare ha dovuto fare i conti con la terza dimensione, affrontando una vera e propria rivoluzione del concetto di genoma nello spazio
25 aprile1953: grazie agli studi di Rosalind Franklin, Watson e Crick propongono un modello che descriva la struttura del DNA. Quattro basi azotate, quattro lettere elegantemente e regolarmente appaiate per formare una meravigliosa doppia elica, capace di conservare l’informazione della vita.
Quattro lettere assemblate in combinazioni diverse per “scrivere” i geni, istruzionifondamentali per la costruzione di ogni pezzo della cellula. Questi vengono letti e “copiati”in RNA, il quale, come un messaggero alato, trasporta l’informazione fino alle officine preposte alla decifrazione e all’assemblaggio delle proteine, effettrici del messaggio.
Ma quanto è lungo questo libretto di istruzioni? Se immaginassimo di srotolare il DNA di una cellula eucariote otterremmo un nastro lungo all’incirca 2 metri di informazione, contenuti in una cellula delle dimensioni di pochi micrometri.
Sembrerebbe un paradosso, ma il segreto sta nell’impacchettamento a cui il DNA va incontro. La sequenza dibasi azotate si arrotola attorno a proteine dette istoni come un filo attorno ad un rocchettoa formare una struttura simile aduna collanadi perle. A loro volta, le perle interagiscono l’una con l’altra, attorcigliandosi e connettendosi sempre di più fino alla forma più compatta di DNA: i cromosomi.
Per anni abbiamo considerato il DNA solo ed esclusivamente nella sua struttura lineare: un semplice susseguirsi di geni letti e copiati indipendentemente l’uno dall’altro. E se proprio avessimo dovuto descrivere il genoma nella sua interezza lo avremmo immaginato come una scatola piena di fili intricati, qualcosa di molto simile a un piatto di spaghetti, ognuno dei quali costituito da una successione di geni trascritti indipendentemente. Negli ultimi anni la biologia molecolare ha dovuto fare i conti con la terza dimensione, affrontando una vera e propria rivoluzione del concetto di genoma nello spazio.
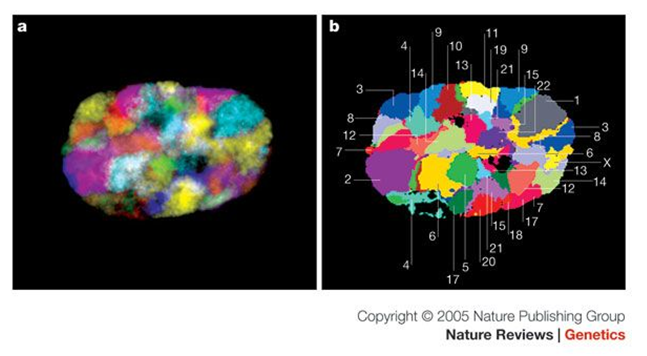
Innanzitutto, il nastro di DNA non è disposto nel nucleo in maniera così confusa e disordinata come un piatto di spaghetti; del resto l’evoluzione non lascerebbe nulla in preda al disordine. Se potessimo salire a bordo di una piccolo navicella ed esplorare l’universo del nucleo come in una vecchia puntata di esplorando il corpo umano, scopriremmo che ogni cromosoma occupa nello spazio un posto ben preciso, chiamato territorio cromosomico. Se, infatti, facciamo una foto al nucleo di una cellula dopo aver colorato ogni cromosoma con un colore diverso, non otteniamo un intreccio di colori (come accadrebbe se il genoma fosse organizzato secondo il modello “a piatto di spaghetti”), ma ogni colore, e quindi ogni cromosoma, occupa una posizione ben precisa nel nucleo.
In questa sorta di paesaggio cromosomico, i cui territori hanno posizioni ben precise a seconda del tipo di cellula in cui ci troviamo, i geni ai confini dei territori vengono letti e trascritti, o per dirla in gergo biologico “espressi”, mentre i geni che si trovano nel cuore del territorio sono in genere silenti.
Nel meraviglioso panorama che è il genoma, il gene non è più un’entità isolata, ma molti geni, presenti su cromosomi diversi,e posizionati alle periferie dei territori cromosomici, interagiscono tra loro nello spazio intercromosomico e vengono trascritti contemporaneamente da proteine chiamate polimerasi all’interno di cosiddette “aziende” di trascrizione. Una visione che richiede un cambio di prospettiva radicale e che spiega perché geni linearmente vicini sullo stesso cromosoma occupino tridimensionalmente posizioni molto lontane, e dunque abbiano profili di espressione molto diversi.
In questa distribuzione spaziale dell’informazione genetica risiede parte della differenza tra un tipo cellulare e l’altro: i geni che si trovano nelle aziende di trascrizione di una cellula del muscolo occuperanno una posizione nello spazio molto diversa nei territori cromosomici di una cellula nervosa, sebbene la sequenza lineare di geni sia esattamente la stessa.
E la regolazione di questa localizzazione diventa ancora più finemente controllata se prendiamo in considerazione la lamina nucleare, quell’intreccio filamentoso di proteine che, come uno steccato, sostengono la membrana nucleare dall’interno. Il DNA che si trova associato alla lamina nucleare è silente e da esso lunghi nastri di DNA si proiettano verso il centro della cellula per essere trascritti.
In una visione tridimensionale tutto acquista un senso, e quella semplice sequenza di quattro lettere si avvolge su se stessa e si condensa, organizzandosi in maniera ordinata e posizionandosi nello spazio a regolare l’espressione genica in maniera molto più raffinata di quanto avremmo potuto pensare.
E cosi, come sempre nella scienza, le cose sono molto più complesse, ma molto più affascinanti di quello che sembrano, e la nostra visione semplicistica e semplificata rende poco merito al meraviglioso tocco dell’evoluzione. E ancora una volta, spettatori attoniti davanti alla meraviglia della natura, ci perdiamo a seguire questi intrecci di geni, maestri di ordine e organizzazione. Tante ancora le domande aperte, tanto ancora da capire su come sianodefiniti e generati i territori cromosomici, come vengano preferite alcune interazioni tra geni invece di altre, che ruolo possa avere lo stato fisico della materia in questa organizzazione spaziale. E noi, a bordo della nostra immaginaria navicella spaziale, continuiamo a viaggiare in questo intreccio meraviglioso che è il nucleo, certi che la soluzione che l’evoluzione ai problemi della vita supera qualsiasi aspettativa umana.
di Lavinia Bisceglie
Questo articolo è stato realizzato per la rubrica Comunicare la scienza, realizzata in collaborazione con gli studenti del Master Cose dell’Università degli studi di Parma
Fonti:
Nature Reviews Genetics 6, 782–792 (2005)
Nature Reviews Genetics volume 8, pages 104–115 (2007)






Scrivi un commento